X线衍射、核磁共振光谱和冷冻电镜是经典的实验性结构生物学研究技术,为无数的生物学问题找到了直接、精确的答案。但这些经典的技术仍然存在方法上的局限性,如均依赖于蛋白表达和纯化,而体外纯化的蛋白和复合体并不一定反映其在细胞内的结构。并且,这些经典方法在解析极难纯化的蛋白复合体和瞬时组装的复合体往往存在困难。近日,在CASP14上拔得头筹的AlphaFold为结构生物学研究注入新的惊喜,但距离AI精确预测结构仍有很远的路要走。
相对于结构生物学家,遗传生物学家青睐于研究遗传互作解析基因间的功能,并利用酵母为生物模型,绘制了大量基因互作图谱。Charles Boone课题组等在2001和2004年分别发表了Science论文工作【1, 2】,发布了SGA(Synthetic Genetic Array)技术,利用该方法研究双突变基因酵母细胞表型,绘制了酵母内的基因互作图谱,解析了多种蛋白的潜在新功能。
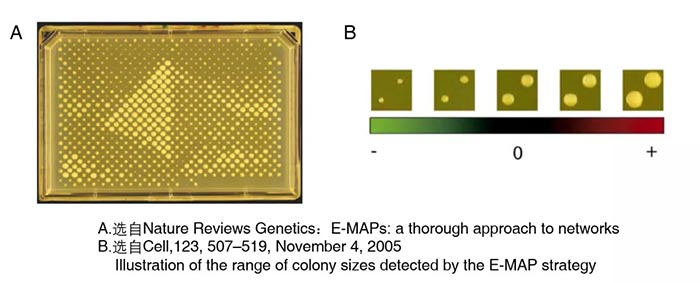
2005年,加州大学旧金山分校的Nevan J. Krogan课题组在Cell上发表了E-MAP方法(Epistatic Miniarray Profiles)【3】,以弥补SGA的不足。SGA用于遗传互作鉴定的表型研究较为有限,主要根据两个基因是否具有合成致死效应来判断互作。E-MAP通过对酵母细胞克隆大小等表型进行定量分析,可以相对预测出基因互作的强度。
在2013年的Cell上,Nevan J. Krogan课题组(Hannes Braberg为第一作者)发表了题为 From Structure to Systems: High-Resolution, Quantitative Genetic Analysis of RNA Polymerase II 的研究,拓展E-MAP为pE-MAP(point mutant epistatic miniarray profile)【4】。通过对RNA聚合酶II 53个点突变进行了6000对定量基因互作分析,解析了RNA聚合酶II亚基之间和其他蛋白复合体间在特定位点间功能上的相关性。这一项工作实现在氨基酸序列上利用基因互作技术分析蛋白结构和功能。
时至今日, Science于12月11日发表了Krogan课题组题为 Genetic interaction mapping informs integrative structure determination of protein complexes 的研究工作。本研究在2013年pE-MAP工作成功的基础上,利用深度定量点突变的基因互作分析解析蛋白结构。

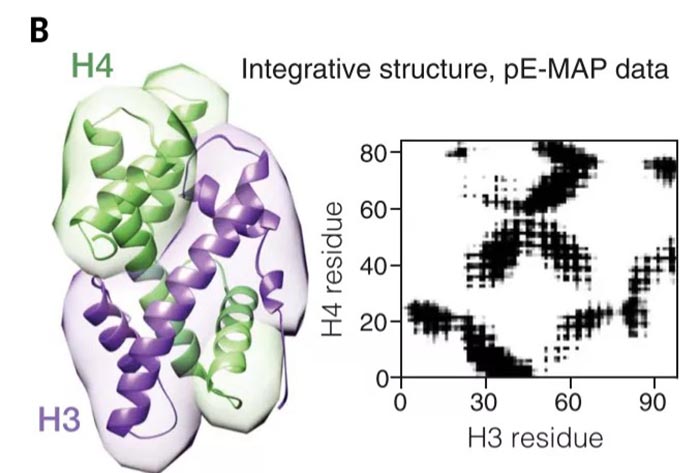
研究人员在本研究中解析了三个结构。其中最主要为酵母组蛋白H3-H4复合体:为分析H3-H4复合体中各位点在结构上的定位,结合深度丙氨酸扫描和修饰残基的特定突变【5】,同时设计了N端序列截短的H3-H4序列,在酵母细胞中实现对内源H3和H4的双基因替换,构建了350株酵母突变细胞株,随后与酵母细胞的1370个基因的敲除或者敲低细胞株进行单倍体配对:

通过对每个酵母单克隆表型进行分析,可以获得不同突变亚克隆与其他基因在功能上的定量分析数据,再通过基因聚类,绘制成该350种突变与其他基因的互作图谱:

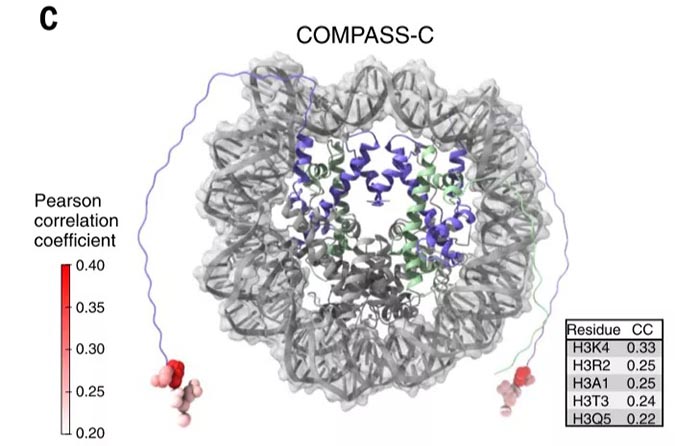
研究人员在数据分析中,设计了MIC (maximal information coefficient)策略进行表型的定量分析。两个邻近的位点比结构上较远的位点具有更高的MIC值。通过对50万对基因著作数据进行综合建模,最后绘制了组蛋白H3-H4的结构。该结构对比已有的经X衍射获得的结构数据具有较高的精确度。研究人员同时也绘制了Histone H3-H4zai COMPASS-C中与其他蛋白的互作结构图。
为确定该方法具有普适性,研究人员以该流程,对酵母RNAPII和细菌RNAP进行了多达6万个基因互作分析,并完成结构解析,并发现该技术可与已有的化学交联和共进化的分析数据相媲美。
在同期的Science杂志上还发表了题为 Using genetics to reveal protein structure 的评论。评论对比了传统的结构生物学方法和以pE-MAP为基础的结构分析,认为pE-MAP可以更真实的反映蛋白复合体在细胞内的原始状态,且该方法不再依赖蛋白纯化的经典的生物化学技术,通过遗传学手段根据功能性实验即可进行结构的定量分析。对于结构存在高度可变和瞬时性的复合结构的解析,pE-MAP具有十足的优势。尽管pE-MAP背后需要巨大的工作量和庞大的数据运算,但随着基因编辑技术和AI深度学习的进步,我们会逐步实现对蛋白复合体“Native”结构的解析。
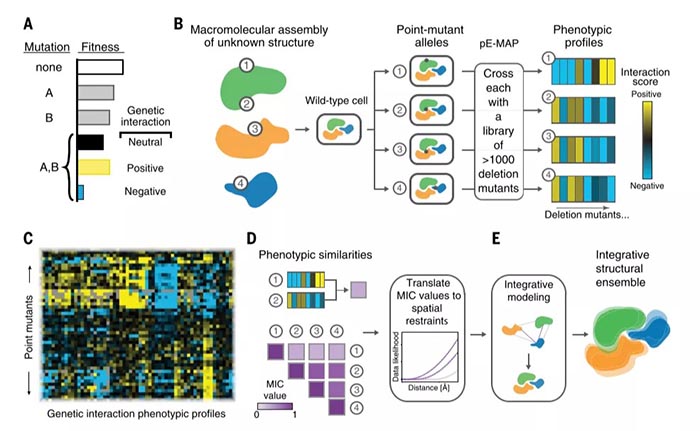
pE-MAP进行结构分析流程图
原文链接:
1. https://science.sciencemag.org/content/370/6522/eaaz4910
2. https://science.sciencemag.org/content/370/6522/1269
参考文献 [1]TONG A H Y, LESAGE G, BADER G D, 等. Global Mapping of the Yeast Genetic Interaction Network[J]. Science (New York, N.Y.), 2004, 303(5659): 808–813. DOI:10.1126/science.1091317. [2]TONG A H, EVANGELISTA M, PARSONS A B, 等. Systematic Genetic Analysis with Ordered Arrays of Yeast Deletion Mutants[J]. Science (New York, N.Y.), 2001, 294(5550): 2364–2368. DOI:10.1126/science.1065810. [3]SCHULDINER M, COLLINS S R, THOMPSON N J, 等. Exploration of the Function and Organization of the Yeast Early Secretory Pathway through an Epistatic Miniarray Profile[J]. Cell, 2005, 123(3): 507–519. DOI:10.1016/j.cell.2005.08.031. [4]BRABERG H, JIN H, MOEHLE E A, 等. From Structure to Systems: High-Resolution, Quantitative Genetic Analysis of RNA Polymerase II[J]. Cell, 2013, 154(4): 775–788. DOI:10.1016/j.cell.2013.07.033. [5]DAI J, HYLAND E M, YUAN D S, 等. Probing Nucleosome Function: A Highly Versatile Library of Synthetic Histone H3 and H4 Mutants[J]. Cell, 2008, 134(6): 1066–1078. DOI:10.1016/j.cell.2008.07.019.













